pandas

公式HP:https://pandas.pydata.org/
例のごとくwikiに頼るとpandasの特徴は
ファイルからデータ読み込んで何か処理したいのであればpandasを使うことになるだろう。 NumPyでもできなくはないのだが、欠損値の処理やグループ化、ピボットテーブルの作成など データの加工を行いたいときはpandasの方がはるかに簡潔に書ける。
- データ操作のための高速で効率的なデータフレーム(DataFrame)オブジェクト
- メモリ内のデータ構造とその他のフォーマットのデータ間で相互に読み書きするためのツール群。フォーマット例: CSV、テキストファイル、Excel、SQLデータベース、HDF5フォーマットなど
- かしこいデータのアライメントおよび統合された欠損値処理
- データセットの柔軟な変形およびピボット
- ラベルに基づいたスライス、fancyインデクシング、巨大なデータセットのサブセット取得
- データセットに対するsplit-apply-combine操作を可能にするエンジンが提供するpowerful groupを使ったデータの集計および変換
- 高性能なデータセットのマージと結合
- 時系列データ: 日付範囲生成、周波数変換、移動窓を用いた統計値や線形回帰、シフトと遅延、など
- パフォーマンスのための高度な最適化。重要なコードはCythonまたはC言語で実装されている。
ネット上にある参考になりそうなサイト一覧
データフレーム (DataFrame) オブジェクト
pandasの扱い方とはDataFrameオブジェクトの扱い方に等しい。
import pandas as pd # pdとすることが多い
# データフレーム作成
data_dict = {'col1': [1, 2, 3], 'col2': [4, 5, 6]}
df1 = pd.DataFrame(data=data_dict) #辞書を渡して作成
print(df1)
下記のような表が出力されるであろう。
| col1 | col2 | |
|---|---|---|
| 0 | 1 | 4 |
| 1 | 2 | 5 |
| 2 | 3 | 6 |
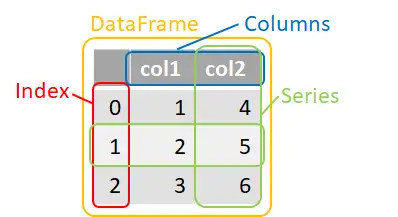
データフレームの構造
"Series"はデータフレーム内の単一の列または行のことを言う。 逆にいうとSeriesが2つ以上集まればDetaFrameである。 Numpyの2次元配列にカラム名やインデックス名が付いたようなものと考えると良いかもしれない。
特定の行または列の取得
下記コードは対話型で一行づつ実行した方が分かりやすいかもしれない。
import pandas as pd
data_dict = {'age': [21, 45, 36], 'point': [46, 34, 29]}
df1 = pd.DataFrame(data=data_dict, index=['A', 'B', 'C']) #Indexを指定
print(df1)
# 列選択
print('--- get Column ---')
print(df1['age']) # [列名]で抽出
print(df1.age) # ドットでも可
# locプロパティ(名前で取得)
print('--- loc ---')
print(df1.loc['A', 'point']) # IndexがAで、列名がpointの要素
# スライス:を用いると複数の要素を取得可能
print('--- loc slice ---')
print(df1.loc['A':'B', 'point']) # IndexがA~Bで、列名がpointの要素
print(df1.loc['A', :]) # IndexがAの行を取得
print(df1.loc[:, 'age']) # df1['age']と同じ
# リストで指定もできる
print('--- loc list ---')
print(df1.loc[['A', 'C'], 'age']) # IndexがAとCで列名がage
# ilocプロパティ(番号で取得)
print('--- iloc ---')
print(df1.iloc[0, 1]) # Index番号が0(=A)で、列番号が1(=point)の要素
# スライス:を用いると複数の要素を取得可能(NumPyの2次元配列と同じ)
print('--- iloc slice ---')
print(df1.iloc[0:2, 1]) # Indexが0~1で(2は含まない)、列番号が1
print(df1.iloc[0, :]) # df1.loc['A', :]と同じ
print(df1.iloc[:, 0]) # df1['age']と同じ
# リストで指定もできる
print('--- iloc list ---')
print(df1.iloc[[0, 2], 0]) # df1.loc[['A', 'C'],'age']と同じ
行または列の追加及び削除
列の追加は容易だが、行の追加はやや面倒である。
import pandas as pd
data_dict = {'age': [21, 45, 36], 'point': [46, 34, 29]}
df1 = pd.DataFrame(data=data_dict, index=['A', 'B', 'C']) #Indexを指定
print(df1)
# 新規列追加
df1['new_col'] = 2 # スカラーを追加
df1['score'] = [78, 84, 68] # リスト追加(ndarrayでも可)
# Seriesの演算とかも可能
df1['calc'] = df1['age'] * df1['point']
print(df1)
# locプロパティでの追加
df1.loc['D'] = [9, 73, 5, 55, 0] # locで新しく行を追加する。
df1.loc[:, 'point2'] = 0 # 新規列の追加
print(df1)
# 削除
df1.drop('D', inplace=True) # inplaceをTrueにすると元のDataFrameが変更される
df1 = df1.drop('new_col', axis=1) # axis=1で列を選択。省略するとaxis=0で行を削除する
print(df1)
なお、追加するにはappend()というのもあるが、 Indexの指定が解除され0,1,…に置き換わったりする。 さらに、速度も遅いのでお勧めしない。
条件に応じて処理する
この当りから記述に慣れがいる。 便利なんですけどね。
import pandas as pd
data_dict = {'age': [21, 45, 36, 30], 'point': [46, 34, 29, 36]}
df1 = pd.DataFrame(data=data_dict)
print(df1)
# pointが40以上ならTrue
print(df1['point'] > 40)
print(df1[df1['point']>40])
# Trueな行だけ選択し、新規列を作成
df1.loc[df1['point'] > 40, 'judge'] = True # スカラーを渡す
df1.loc[df1['point'] < 40, 'point2'] = df1['point'] * 1.5
print(df1)
# 既存の列に適用
df1.loc[df1['point'] > 40, 'point2'] = df1['point']
print(df1)
条件に応じた処理にはwhere()やmask()メソッドというものある。
where() / mask()メソッド
where()は条件に対してFalseのものをNaNまたは第2引数の値にする。
import pandas as pd
data_dict = {
'name': ['John', 'Tom', 'Alice', 'Emma', 'Alex'],
'age': [20, 13, 25, 17, 15],
'score': [67, 55, 74, 60, 62]
}
df1 = pd.DataFrame(data=data_dict)
df1['class'] = 'A'
print(df1.where(df1['age'] > 17))
print(df1['score'].where(df1['age']>17, df1['score']*1.2))
df1['score2'] = df1['score'].where(df1['age']>17, df1['score']*1.2)
# 元のオブジェクトを変更したければ、inplace=True
df1['class'].where(df1['score2']>70, 'B', inplace=True)
print(df1)
mask()は条件に対してTrueのものをNaNまたは、第2引数の値にする。
import pandas as pd
data_dict = {
'name': ['John', 'Tom', 'Alice', 'Emma', 'Alex'],
'age': [20, 13, 25, 17, 15],
'score': [67, 55, 74, 60, 62]
}
df1 = pd.DataFrame(data=data_dict)
df1['class'] = 'A'
print(df1.mask(df1['age'] > 17))
print(df1['score'].mask(df1['age']>17, df1['score']*1.2))
df1['score2'] = df1['score'].mask(df1['age']>17, df1['score']*1.2)
# 元のオブジェクトを変更したければ、inplace=True
df1['class'].mask(df1['score2']>70, 'B', inplace=True)
print(df1)
pandasのwhereおよびmaskメソッドでは、 第2引数にTrueまたはFalseのときの変更後の値を設定できるが、 もう一方は元のオブジェクトの値がそのまま使われる。 Numpyにもwhereメソッドが存在し、 こちらは第1引数に条件、第2引数にTrueの時の値、第3引数にFlaseの時の値を渡すことが出来る。
import pandas as pd
import numpy as np
data_dict = {
'name': ['John', 'Tom', 'Alice', 'Emma', 'Alex'],
'age': [20, 13, 25, 17, 15],
'score': [67, 55, 74, 60, 62]
}
df1 = pd.DataFrame(data=data_dict)
df1['score2'] = df1['score'].where(df1['age']>17, df1['score']*1.2)
df1['class'] = np.mask(df1['score2']>70, 'A', 'B')
print(df1)
ファイルの読み込みと統計処理
「とりあえずcsv読み込むならpandasで」とよく書かれている。それぐらい便利。
import pandas as pd
# 特に指定せずに読み込んだ場合
df = pd.read_csv('sample_data2.txt', sep='\t')
print(df)
print(df.dtypes) # pandasが賢いので科目列はintとして読み込んでくれる
# いろいろ指定して読み込むこともできる
COL_TYPES = {'氏名':str, 'クラス':str, '英語':int, '数学':int, '国語':int, '理科':int,'社会':int}
df = pd.read_csv(
'sample_data2.txt',
sep = '\t',
dtype = COL_TYPES, # 列ごとに型を明示
index_col = 0, # Indexとして利用する列を指定
encoding = 'UTF-8' # 文字コード指定
)
print(df.head()) # 最初の5行
# 統計量(数値型以外の列は無視してくれる)
print(df.mean()) # 平均
print(df.std()) # 標準偏差
print(df.describe()) # いろいろまとめて計算
# 列の追加
df['tot'] = df.sum(axis=1) # 行ごとの合計
df['ave'] = df.mean(axis=1) # 行ごとの平均
print(df.head())
# ソート
print(df.sort_values('tot', ascending=False))
何かしらの計算を行うときに数値型かどうかを自動で判定し、処理を行う列をpandasが決めてくれる。
データの加工(グループ化やpivotテーブル)
NumPyでは面倒であったグループ化の処理も用意にできる。
import pandas as pd
# いろいろ指定してやる
COL_TYPES = {'氏名':str, 'クラス':str, '英語':int, '数学':int, '国語':int, '理科':int,'社会':int}
df = pd.read_csv('sample_data2.txt', sep='\t', dtype=COL_TYPES, index_col=0, encoding='UTF-8')
# クラスごとにグループ化
df_groupby_class = df.groupby('クラス')
print(df_groupby_class.mean()) # 平均計算
pandasはdatetime(日付)に関する処理も強力なので以下にその例を示す。 用いているサンプルデータは都道府県別のCOVID-19の感染者数データ (出典:NHK) で
| 日付 | 都道府県コード | 都道府県名 | 各地の感染者数_1日ごとの発表数 | 各地の感染者数_累計 | 各地の死者数_1日ごとの発表数 | 各地の死者数_累計 | 各地の直近1週間の人口10万人あたりの感染者数 |
|---|---|---|---|---|---|---|---|
| 2020/1/16 | 01 | 北海道 | 0 | 0 | 0 | 0 | |
| ・ | ・ | ・ | ・ | ・ | ・ | ・ | ・ |
| ・ | ・ | ・ | ・ | ・ | ・ | ・ | ・ |
import pandas as pd
df = pd.read_csv(
'sampledata_covid19_220303.csv',
sep = ',',
parse_dates = ['日付'],
encoding = 'UTF-8')
print(df.dtypes)
# 使用する列選択
df = df.loc[:, ['日付', '都道府県名','各地の感染者数_1日ごとの発表数', '各地の死者数_1日ごとの発表数']]
# 列名が長いので変更
re_columns = {
'日付': 'date',
'都道府県名': 'prefecture',
'各地の感染者数_1日ごとの発表数': 'infections',
'各地の死者数_1日ごとの発表数': 'death'
}
df.rename(columns=re_columns, inplace=True)
print(df)
# pivotテーブル作成(crosstab()というものある)
df_pivot = df.pivot_table(index='date', columns='prefecture', values=['infections', 'death'])
print(df_pivot)
# 1ヶ月毎に集計
df_pivot_month = df.pivot_table(
index = pd.Grouper(key='date', freq='M'), # Mで1ヶ月、2Mで2ヶ月、7Dで7日など指定出来る
columns = 'prefecture',
values = ['infections', 'death'],
aggfunc = sum # 1ヶ月ごとの和を計算
)
print(df_pivot_month)
# 保存
#df_pivot_month.to_csv('ivot_month_covid19_220303.csv') # csv形式で出力
df_pivot_month.to_pickle('pivot_month_covid19_220303.pkl') # pkl形式で出力
上記コードにおいてピポットテーブルを作成しているのは24行目と28行目である。 "df_pivot"は一日ごとの集計であり、df_pivot_monthは1ヶ月ごとの集計となる。 日付のグループ化にはpd.Grouper(key, freq)を用いることが多い。 keyには使用する列、freqには集計間隔('D'、'M'以外にも週ごとを表す'W'などもある)を渡す。
また、保存処理もここで紹介しておく。 to_csv()はその名の通りcsv形式で保存するメソッドである。 csv形式への出力は、出力後のデータをエクセルなど別のアプリケーションにいれて処理し直す 場合には便利だが、Python(pandas)で再度読み込む場合には、read_csv()で色々指定し直す 必要があり面倒である。 そのような時に使われるのがto_pickleでこれはDataFrameオブジェクトをそのままバイナリファイルとして 保存する。 csv形式のように人間に読めるデータではない(テキストエディタで開けばおそらく文字化けする)が、 Pythonにとっては直接読めるので処理速度が速くなるだけでなく、 csvと比べてファイルサイズも削減できる(場合もある)。 なお、読み込む際はpd.read_pickle(path)で読み込める。
可視化(matplotlibと兼用)
前のセクションで作った'pivot_month_covid19_220303.pkl'を利用して グラフを作ってみる。
import pandas as pd
import matplotlib.pyplot as plt
import datetime as dt
df = pd.read_pickle('pivot_month_covid19_220303.pkl')
print(df)
# 何も考えずにただplotするなら以下で済む
#df.plot()
#plt.show()
# 東京の新規感染者数
df['infections']['東京都'].plot(label='TOKYO')
df['infections'].mean(axis=1).plot(label='National Ave')
plt.legend()
plt.show()
# 東京・神奈川・埼玉のデータ抽出
df_inf_tks = df['infections'].loc[:, ['東京都','神奈川県','埼玉県']]
df_inf_tks_ave = df_inf_tks.mean(axis=1) # 3都県平均
df_inf_all_ave = df['infections'].mean(axis=1) # 全国平均
# axオブジェクトを作ってからその中にいれることもできる
fig = plt.figure()
ax1 = fig.add_subplot(1, 2, 1)
df_inf_tks.plot(ax=ax1)
df_inf_tks_ave.plot(label='3都県平均', ls='--', ax=ax1)
df_inf_all_ave.plot(label='全国平均', ls='--', c='k', ax=ax1)
ax1.set_title('New Infections')
xmin, xmax = dt.date(2020, 1, 1), dt.date(2022, 2, 28) # x軸範囲
ax1.set_xlim([xmin, xmax])
ax1.legend()
ax2 = fig.add_subplot(1, 2, 2)
df_inf_tks.cumsum().plot(ax=ax2) # cumsumで累計計算
df_inf_tks_ave.cumsum().plot(label='3都県平均', ls='--', ax=ax2)
df_inf_all_ave.cumsum().plot(label='全国平均', ls='--', c='k', ax=ax2)
ax2.set_title('Cumulative Infections')
ax2.set_xlim([xmin, xmax])
ax2.legend()
plt.show()
線グラフplot()以外にもdf.plot.bar() [df.plot(kind='bar')]や df.plot.scatter() [df.plot(kind='scatter')]など他のグラフも書ける。 なお、累計を求める際にcumsum()を用いているが、安全に行うならdateをキーにして ソートしてからcumsum()した方がよいだろう。
matplotlibとの連携
matplotlibとの連携をもう少し詳しく見ていく。
DataFrame.plot()
pandasはmatplotlibとの連携が容易にできる。
import pandas as pd
import matplotlib.pyplot as plt
data_dict = {
'x':[1,2,3,5,6,7,9],
'y1':[2,5,4,3,7,9,2],
'y2':[3,4,6,9,2,1,5],
'y3':[1,4,6,9,11,13,15],
}
# DataFrame作成
df = pd.DataFrame(data_dict)
# plot
df.plot()
# 表示
plt.show()
plotを行っているのは13行目である。 plot()に渡す引数を省略した場合、x軸はdfのIndexの値となり、 dfが持つ['x', 'y1', 'y2', 'y3']列の全てyの値として描画される。 plotに渡せる引数は主に以下である。 (大量にあるので詳しくは公式を確認すると良い。)
df.plot(
data # DataFrameかSeries
x # labelかposition
y # labelかposition
kind # グラフの種類
ax # axオブジェクト
)
xに'x'列を指定し、yを'y1'と'y2'に限定する。 また、kindを省略するとデフォルトの'line'が選択されるが、 これを明示してみると
import pandas as pd
import matplotlib.pyplot as plt
data_dict = {
'x':[1,2,3,5,6,7,9],
'y1':[2,5,4,3,7,9,2],
'y2':[3,4,6,9,2,1,5],
'y3':[1,4,6,9,11,13,15],
}
# DataFrame作成
df = pd.DataFrame(data_dict)
# plot
df.plot(x='x', y=['y1','y2'], kind='line')
# 表示
plt.show()
kindに'line'を指定したがこれは別の書き方も可能で、
df.plot.line()
と同じ結果になる。また、kindで指定できるグラフは以下である。
| 引数名 | グラフ種類 |
|---|---|
| line | 線グラフ(デフォルト) |
| bar | 棒グラフ(鉛直方向) |
| barh | 棒グラフ(水平方向) |
| hist | ヒストグラム |
| box | 箱ひげ図 |
| kde | カーネル密度推定 |
| density | カーネル密度推定のようなもの |
| area | 面グラフ |
| pie | 円グラフ |
| scatter | 散布図 |
| hexbin | 2次元ヒストグラム |
引数axには独自に用意したaxオブジェクト渡すことができる。 DataFrame.plot()に渡せる引数の中にはグラフの細かい設定を行うための引数が容易されているのだが、 渡す引数の数が多くなり、可読性が悪くなる。 そこで、axオブジェクトを別に用意し、それを調整したものをDataFrame.plot()に渡した方が分かりやすい。 axオブジェクトはmatplotlibのものであるから、matplotlibで扱った時と同じ扱いでよい。
import pandas as pd
import matplotlib.pyplot as plt
data_dict = {
'x':[1,2,3,5,6,7,9],
'y1':[2,5,4,3,7,9,2],
'y2':[3,4,6,9,2,1,5],
'y3':[1,4,6,9,11,13,15],
}
# DataFrame作成
df = pd.DataFrame(data_dict)
'''
# plot
df.plot.line(
x = 'x',
xlim = [1, 8],
ylim = [0, 16],
grid=True,
title='test',
figsize=(5, 5)
)
'''
fig, ax = plt.subplots(figsize=(5, 5))
df.plot.line(x='x', ax=ax)
ax.set_xlim(1, 8)
ax.set_ylim(0, 16)
ax.grid()
ax.set_title('test')
# 表示
plt.show()
コメントアウトした14~20行目でplot()の引数に色々渡して設定するか、 23行目に書いたようにaxオブジェクトを用意し、それを27~30行目のように設定するか という違いになる。
実際は別の書き方もできて、DataFrame.plot()はaxオブジェクトを返すので、 その返されたaxオブジェクトに対して設定を行っても良い。
import pandas as pd
import matplotlib.pyplot as plt
data_dict = {
'x':[1,2,3,5,6,7,9],
'y1':[2,5,4,3,7,9,2],
'y2':[3,4,6,9,2,1,5],
'y3':[1,4,6,9,11,13,15],
}
# DataFrame作成
df = pd.DataFrame(data_dict)
# plot
ax = df.plot.line(x='x')
ax.set_xlim(1, 8)
ax.set_ylim(0, 16)
ax.grid()
ax.set_title('test')
# 表示
plt.show()
個人的なおすすめは2番目に紹介したfigureとaxオブジェクトを別に用意する方法である。 その理由としてはmatplotlibと同じように設定すれば済む (複雑なグラフのレイアウト設定などもmatplotlibでやった様に書ける)という点と、 オブジェクトの生成を明示した方が読みやすい気がするからである。
axオブジェクトを生成することで、グラフを重ねることも容易になる。
import pandas as pd
import matplotlib.pyplot as plt
data_dict1 = {
'x':[1,2,3,5,6,7,9],
'y1':[2,5,4,3,7,9,2],
'y2':[3,4,6,9,2,1,5],
'y3':[1,4,6,9,11,13,15],
}
data_dict2 = {
'X':[2, 4, 6, 7, 8],
}
# DataFrame作成
df1 = pd.DataFrame(data_dict1)
df2 = pd.DataFrame(data_dict2)
df2['Y1'] = df2['X'] ** (1/2)
fig, ax = plt.subplots(figsize=(5, 5))
df1.plot.line(x='x', ax=ax)
df2.plot.scatter(x='X', y='Y1', color='red', marker='D', ax=ax)
ax.set_xlim(1, 8)
ax.set_ylim(0, 16)
ax.grid()
ax.set_title('test')
# 表示
plt.show()
ax.plot()
ここまで紹介したのはDataFrameオブジェクトが持つplot()メソッドを用いたグラフ作成であった。 この方法でも良いのだが、 matplotlibのaxオブジェクトにもplot()メソッドがあったことを思い出して欲しい。
import matplotlib.pyplot as plt
x = [1, 2, 3, 5, 6, 7, 9]
y = [2, 5, 4, 3, 7, 9, 2]
fig, ax = plt.subplots(figsize=(5, 5))
ax.plot(x, y, label='y')
ax.set_xlim(1, 8)
ax.set_ylim(0, 16)
ax.grid()
ax.legend()
ax.set_title('test')
# 表示
plt.show()
結局のところ、ax.plot(x, y)のxとyにDataFrameのSeriesを渡しても同じ挙動になる。 (PandasのSeriesはNumpyのndarrayと同じように扱えるので、plot()の引数に問題なく渡せる。)
import pandas as pd
import matplotlib.pyplot as plt
data_dict = {
'x':[1,2,3,5,6,7,9],
'y1':[2,5,4,3,7,9,2],
'y2':[3,4,6,9,2,1,5],
'y3':[1,4,6,9,11,13,15],
}
# DataFrame作成
df = pd.DataFrame(data_dict)
# plot
fig, ax = plt.subplots(figsize=(5, 5))
print(df.columns)
ax.plot(df['x'], df['y1'], label='y1')
ax.plot(df['x'], df['y2'], label='y2')
ax.plot(df['x'], df['y3'], label='y3')
ax.set_xlim(1, 8)
ax.set_ylim(0, 16)
ax.grid()
ax.legend()
ax.set_title('test')
# 表示
plt.show()
上記例だと、ax.plot()を3回も書いていて面倒なのでplot()部分をfor文で回すようにすると
import pandas as pd
import matplotlib.pyplot as plt
data_dict = {
'x':[1,2,3,5,6,7,9],
'y1':[2,5,4,3,7,9,2],
'y2':[3,4,6,9,2,1,5],
'y3':[1,4,6,9,11,13,15],
}
# DataFrame作成
df = pd.DataFrame(data_dict)
print('オリジナルのdf')
print(df)
print('列リスト', df.columns)
df.index = df['x'] # Indexをx列と同じ値に設定
df.drop('x', axis=1, inplace=True) # x列削除
print('Index設定後')
print(df)
print('列リスト', df.columns)
# figureとax作成
fig, ax = plt.subplots(figsize=(5, 5))
# 列に対してforループ
for col in df.columns:
ax.plot(df.index, df[col], label=col)
ax.set_xlim(1, 8)
ax.set_ylim(0, 16)
ax.grid()
ax.legend()
ax.set_title('test')
# 表示
plt.show()
最終的に言えるのは、グラフ描画するには色々な方法があるので、好きなもの使えばよいということである。